- Seite 1: Quantitative Proteomanalyse von Pse
- Seite 5: Veröffentlichungen Ein Teil der Er
- Seite 8 und 9: Inhaltsverzeichnis 4.2.3 Kultivieru
- Seite 10 und 11: Inhaltsverzeichnis 5.3.2 Analyse de
- Seite 12 und 13: Abbildungsverzeichnis 3.1 Oxidation
- Seite 14 und 15: Abbildungsverzeichnis 5.39 Einteilu
- Seite 16 und 17: Tabellenverzeichnis 5.16 Differenti
- Seite 18 und 19: 1 Zusammenfassung Induktion der Fet
- Seite 20 und 21: 2 Summary The main focus of this wo
- Seite 22 und 23: 3 Einleitung 3.1 Systembiologie in
- Seite 24 und 25: 3 Einleitung 1948). Beide Autoren b
- Seite 26 und 27: 3 Einleitung schritt im Bereich der
- Seite 28 und 29: 3 Einleitung Abbildung 3.2: Glyoxyl
- Seite 30 und 31: 3 Einleitung verschiedener Mechanis
- Seite 32 und 33: 3 Einleitung Pyruvat zu Acetaldehyd
- Seite 34 und 35: 3 Einleitung Wie auch bei der Herst
- Seite 36 und 37: 3 Einleitung Pseudomonas putida KT2
- Seite 38 und 39: 3 Einleitung schwierig. Substanzen,
- Seite 40 und 41: 3 Einleitung werden die Zeit und da
- Seite 42 und 43: 3 Einleitung definierten Massenbere
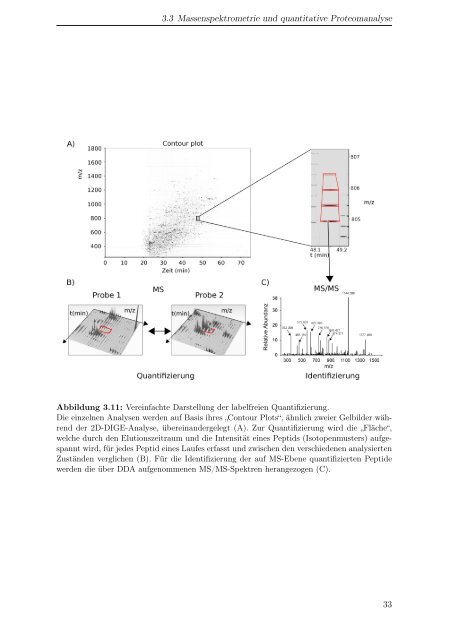
- Seite 44 und 45: 3 Einleitung 3.3.4 Proteinidentifiz
- Seite 46 und 47: 3 Einleitung Silberfärbung überle
- Seite 50 und 51: 3 Einleitung 3.3.8 Selected reactio
- Seite 52 und 53: 3 Einleitung weiter zu erhöhen, is
- Seite 54 und 55: 4 Material und Methoden 4.1 Verwend
- Seite 56 und 57: 4 Material und Methoden Tabelle 4.3
- Seite 58 und 59: 4 Material und Methoden Bezeichnung
- Seite 60 und 61: 4 Material und Methoden Medium Zusa
- Seite 62 und 63: 4 Material und Methoden geführten
- Seite 64 und 65: 4 Material und Methoden 0,05% Oktan
- Seite 66 und 67: 4 Material und Methoden in ein neue
- Seite 68 und 69: 4 Material und Methoden die in eine
- Seite 70 und 71: 4 Material und Methoden 30 min im D
- Seite 72 und 73: 4 Material und Methoden der unteren
- Seite 74 und 75: 4 Material und Methoden Quotienten
- Seite 76 und 77: 4 Material und Methoden 4.5.2 Ident
- Seite 78 und 79: 4 Material und Methoden Tabelle 4.1
- Seite 80 und 81: 4 Material und Methoden 4.5.3.1 bes
- Seite 82 und 83: 4 Material und Methoden Abbildung 4
- Seite 84 und 85: 4 Material und Methoden SRM-Überga
- Seite 86 und 87: 4 Material und Methoden getrennt. D
- Seite 88 und 89: 5 Ergebnisse Abbildung 5.1: Durchf
- Seite 90 und 91: 5 Ergebnisse pH-Gradienten lediglic
- Seite 92 und 93: 5 Ergebnisse Differenzielle Regulat
- Seite 94 und 95: 5 Ergebnisse (31-fach), bei welchem
- Seite 96 und 97: 5 Ergebnisse Position der beiden Sp
- Seite 98 und 99:
5 Ergebnisse Differenzielle Regulat
- Seite 100 und 101:
5 Ergebnisse Für die quantitative
- Seite 102 und 103:
5 Ergebnisse Porin (PP_2662) und ei
- Seite 104 und 105:
5 Ergebnisse Abbildung 5.4: Einteil
- Seite 106 und 107:
5 Ergebnisse Abbildung 5.5: Einteil
- Seite 108 und 109:
5 Ergebnisse Abbildung 5.6: Postuli
- Seite 110 und 111:
5 Ergebnisse Abbildung 5.7: Unter G
- Seite 112 und 113:
5 Ergebnisse 5.2 Untersuchungen zum
- Seite 114 und 115:
5 Ergebnisse ausgesetzt. Grundlage
- Seite 116 und 117:
5 Ergebnisse vorherigen Experiment
- Seite 118 und 119:
5 Ergebnisse Locus-Tag Gen Protein
- Seite 120 und 121:
5 Ergebnisse mungskettenenzym Cytro
- Seite 122 und 123:
5 Ergebnisse nicht bekannt. Auch in
- Seite 124 und 125:
5 Ergebnisse 5.2.2 Transkriptom- un
- Seite 126 und 127:
5 Ergebnisse Abbildung 5.12: VENN-D
- Seite 128 und 129:
5 Ergebnisse Da nicht alle Kultivie
- Seite 130 und 131:
5 Ergebnisse nisse der GeLCMSMS-Ana
- Seite 132 und 133:
5 Ergebnisse wicht des nach der Zel
- Seite 134 und 135:
5 Ergebnisse Abbildung 5.16: Prozen
- Seite 136 und 137:
5 Ergebnisse Abbildung 5.17: Darste
- Seite 138 und 139:
5 Ergebnisse Viele der differentiel
- Seite 140 und 141:
5 Ergebnisse Abbildung 5.19: Regula
- Seite 142 und 143:
5 Ergebnisse Abbildung 5.20: Differ
- Seite 144 und 145:
5 Ergebnisse Locus-Tag Gen Protein
- Seite 146 und 147:
5 Ergebnisse Tabelle 5.19: In den v
- Seite 148 und 149:
5 Ergebnisse Tabelle 5.20: In den v
- Seite 150 und 151:
5 Ergebnisse exprimiert. Darunter d
- Seite 152 und 153:
5 Ergebnisse Abbildung 5.22: Differ
- Seite 154 und 155:
5 Ergebnisse Locus-Tag Gen Protein
- Seite 156 und 157:
5 Ergebnisse Tabelle 5.25: In den v
- Seite 158 und 159:
5 Ergebnisse 5.3.1 Analyse der zyto
- Seite 160 und 161:
5 Ergebnisse Tabelle 5.27: Übersic
- Seite 162 und 163:
5 Ergebnisse Tabelle 5.28: Übersic
- Seite 164 und 165:
5 Ergebnisse nillin induzierten Pro
- Seite 166 und 167:
5 Ergebnisse Tabelle 5.29: Übersic
- Seite 168 und 169:
5 Ergebnisse unter Glucose spricht
- Seite 170 und 171:
5 Ergebnisse Locus-Tag Gen Protein
- Seite 172 und 173:
5 Ergebnisse KT2440 zu. Die Tatsach
- Seite 174 und 175:
5 Ergebnisse PP_3739 zeigte bei ein
- Seite 176 und 177:
5 Ergebnisse portsystems identifizi
- Seite 178 und 179:
5 Ergebnisse Locus-Tag Gen Protein
- Seite 180 und 181:
5 Ergebnisse Abbildung 5.29: Differ
- Seite 182 und 183:
5 Ergebnisse Locus-Tag Gen Protein
- Seite 184 und 185:
5 Ergebnisse Tabelle 5.36: Vergleic
- Seite 186 und 187:
5 Ergebnisse Lipid-Metabolismus Im
- Seite 188 und 189:
5 Ergebnisse Tabelle 5.38: Übersic
- Seite 190 und 191:
5 Ergebnisse in unseren Experimente
- Seite 192 und 193:
5 Ergebnisse Abbildung 5.32: Quanti
- Seite 194 und 195:
5 Ergebnisse mente wurden anschlie
- Seite 196 und 197:
5 Ergebnisse Abbildung 5.34: Vertei
- Seite 198 und 199:
5 Ergebnisse sion verstärkt. So ko
- Seite 200 und 201:
5 Ergebnisse Abbildung 5.37: Differ
- Seite 202 und 203:
5 Ergebnisse Abbildung 5.39: Eintei
- Seite 204 und 205:
5 Ergebnisse gulation von LiuA war
- Seite 206 und 207:
5 Ergebnisse Locus- Gen Protein Glu
- Seite 208 und 209:
5 Ergebnisse Abbildung 5.41: Schema
- Seite 210 und 211:
5 Ergebnisse Tabelle 5.43: Differen
- Seite 212 und 213:
5 Ergebnisse Abbildung 5.42: Gelbil
- Seite 214 und 215:
5 Ergebnisse Tabelle 5.44: Vergleic
- Seite 216 und 217:
5 Ergebnisse Spot gi Nummer Protein
- Seite 218 und 219:
6 Diskussion in P. putida JM37 deut
- Seite 220 und 221:
6 Diskussion al., 1995; Toyama et a
- Seite 222 und 223:
6 Diskussion stoffquelle abhängig
- Seite 224 und 225:
6 Diskussion angeimpft. Die Zugabe
- Seite 226 und 227:
6 Diskussion 3 g/l Butanol könnte
- Seite 228 und 229:
6 Diskussion cken sich dabei nicht
- Seite 230 und 231:
6 Diskussion setzungsversuche von Y
- Seite 232 und 233:
6 Diskussion lin zuließ, war die v
- Seite 234 und 235:
6 Diskussion Analyse unterschiedlic
- Seite 236 und 237:
6 Diskussion mäßig geringen Grö
- Seite 238 und 239:
6 Diskussion Publikation Verwendete
- Seite 240 und 241:
6 Diskussion a) Stabilisierung und
- Seite 242 und 243:
6 Diskussion nicht nachgewiesen wer
- Seite 244 und 245:
6 Diskussion senspektrometrischer Q
- Seite 246 und 247:
6 Diskussion Anteil an Proteinen de
- Seite 248 und 249:
6 Diskussion gezielte und absolute
- Seite 250 und 251:
6 Diskussion Wie bereits in den üb
- Seite 252 und 253:
Literatur Alban, Andrew, Stephen Ol
- Seite 254 und 255:
Literatur Chattopadhyay, Ava, Karin
- Seite 256 und 257:
Literatur bp inverted repeat sequen
- Seite 258 und 259:
Literatur Inui, Masayuki, Masako Su
- Seite 260 und 261:
Literatur Lovric, Josip (2011). Int
- Seite 262 und 263:
Literatur comparative analysis of t
- Seite 264 und 265:
Literatur coding three quinoprotein
- Seite 266 und 267:
Literatur Schmidt, Alexander, Josef
- Seite 268 und 269:
Literatur Toyama, Hirohide, Zhi-Wei
- Seite 270 und 271:
Literatur Wood J. M., W. A. und R.
- Seite 272 und 273:
Abkürzungsverzeichnis CE DC GeLCMS
- Seite 274 und 275:
Eidesstattliche Versicherung Eidess
- Seite 276:
Anhang Alle Tabellen und Abbildunge