Protein ? Disassembly im Verlauf der endosomalen Prozessierung
Protein ? Disassembly im Verlauf der endosomalen Prozessierung
Protein ? Disassembly im Verlauf der endosomalen Prozessierung
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Ergebnisse Seite 104<br />
MHC Klasse II- und DM Röntgen - Strukturdaten liegen sowohl von humanen als auch<br />
von murinen Molekülen in unterschiedlichen Auflösungen vor. Einen Überblick über die<br />
vorhandenen Strukturen gibt Tab. 15<br />
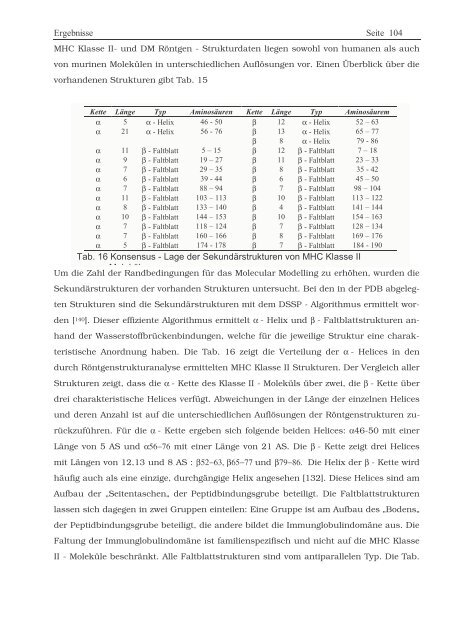
Kette Länge Typ Aminosäuren Kette Länge Typ Aminosäurem<br />
α 5 α - Helix 46-50 β 12 α - Helix 52–63<br />
α 21 α - Helix 56-76 β 13 α - Helix 65–77<br />
β 8 α- Helix 79-86<br />
α 11 β - Faltblatt 5–15 β 12 β - Faltblatt 7–18<br />
α 9 β- Faltblatt 19–27 β 11 β - Faltblatt 23–33<br />
α 7 β- Faltblatt 29–35 β 8 β- Faltblatt 35-42<br />
α 6 β- Faltblatt 39-44 β 6 β- Faltblatt 45–50<br />
α 7 β- Faltblatt 88–94 β 7 β- Faltblatt 98 – 104<br />
α 11 β - Faltblatt 103 – 113 β 10 β - Faltblatt 113 – 122<br />
α 8 β - Faltblatt 133 – 140 β 4 β - Faltblatt 141 – 144<br />
α 10 β - Faltblatt 144 – 153 β 10 β - Faltblatt 154 – 163<br />
α 7 β - Faltblatt 118 – 124 β 7 β - Faltblatt 128 – 134<br />
α 7 β - Faltblatt 160 – 166 β 8 β - Faltblatt 169 – 176<br />
α 5 β - Faltblatt 174 - 178 β 7 β - Faltblatt 184 - 190<br />
Tab. 16 Konsensus - Lage <strong>der</strong> Sekundärstrukturen von MHC Klasse II<br />
M l kül<br />
Um die Zahl <strong>der</strong> Randbedingungen für das Molecular Modelling zu erhöhen, wurden die<br />
Sekundärstrukturen <strong>der</strong> vorhanden Strukturen untersucht. Bei den in <strong>der</strong> PDB abgeleg-<br />
ten Strukturen sind die Sekundärstrukturen mit dem DSSP - Algorithmus ermittelt wor-<br />
den [ 140]. Dieser effiziente Algorithmus ermittelt α - Helix und β - Faltblattstrukturen an-<br />
hand <strong>der</strong> Wasserstoffbrückenbindungen, welche für die jeweilige Struktur eine charak-<br />
teristische Anordnung haben. Die Tab. 16 zeigt die Verteilung <strong>der</strong> α - Helices in den<br />
durch Röntgenstrukturanalyse ermittelten MHC Klasse II Strukturen. Der Vergleich aller<br />
Strukturen zeigt, dass die α - Kette des Klasse II - Moleküls über zwei, die β - Kette über<br />
drei charakteristische Helices verfügt. Abweichungen in <strong>der</strong> Länge <strong>der</strong> einzelnen Helices<br />
und <strong>der</strong>en Anzahl ist auf die unterschiedlichen Auflösungen <strong>der</strong> Röntgenstrukturen zu-<br />
rückzuführen. Für die α - Kette ergeben sich folgende beiden Helices: α46-50 mit einer<br />
Länge von 5 AS und α56−76 mit einer Länge von 21 AS. Die β - Kette zeigt drei Helices<br />
mit Längen von 12,13 und 8 AS : β52−63, β65−77 und β79−86. Die Helix <strong>der</strong> β - Kette wird<br />
häufig auch als eine einzige, durchgängige Helix angesehen [132]. Diese Helices sind am<br />
Aufbau <strong>der</strong> „Seitentaschen„ <strong>der</strong> Peptidbindungsgrube beteiligt. Die Faltblattstrukturen<br />
lassen sich dagegen in zwei Gruppen einteilen: Eine Gruppe ist am Aufbau des „Bodens„<br />
<strong>der</strong> Peptidbindungsgrube beteiligt, die an<strong>der</strong>e bildet die Immunglobulindomäne aus. Die<br />
Faltung <strong>der</strong> Immunglobulindomäne ist familienspezifisch und nicht auf die MHC Klasse<br />
II - Moleküle beschränkt. Alle Faltblattstrukturen sind vom antiparallelen Typ. Die Tab.