Tagungsband - UFZ
Tagungsband - UFZ
Tagungsband - UFZ
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
<strong>Tagungsband</strong> Statusseminar des BMBF-Ad-hoc-Verbundprojektes in Freiberg, 27.-29.08.2003<br />
propagiert, welche mit dem Flutwasser in Berührung gekommen waren. Wo diese Desinfektion<br />
vorgenommen wurde, wurden von uns keine Proben genommen, um den Datensatz nicht zu<br />
divers zu machen. Die Proben werden vor Ort fixiert bzw. eingefroren, um eine nachträgliche<br />
Veränderung der mikrobiellen Gemeinschaften zu verhindern. Der Teil der Proben, welcher für<br />
die Kultivierung auf Selektivagar-Nährböden vorgesehen war, wurde unmittelbar nach der<br />
Rückkehr ins Labor ausplattiert. Die Agarnährböden wurden nach den EU-Richtlinien für<br />
Gewässerhygiene kultiviert und die entstehenden Kolonien wurden ausgezählt, wobei nach<br />
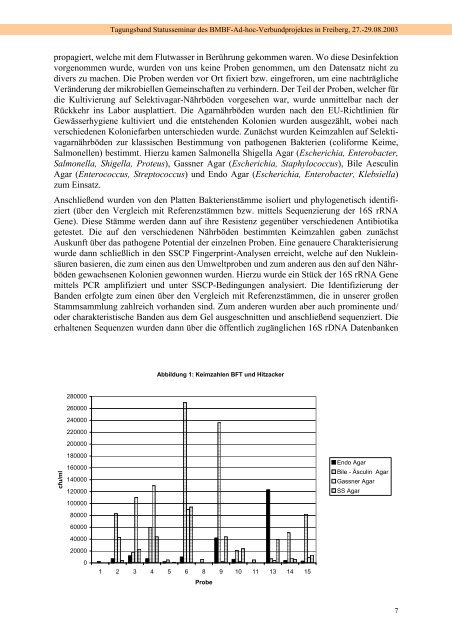
verschiedenen Koloniefarben unterschieden wurde. Zunächst wurden Keimzahlen auf Selektivagarnährböden<br />
zur klassischen Bestimmung von pathogenen Bakterien (coliforme Keime,<br />
Salmonellen) bestimmt. Hierzu kamen Salmonella Shigella Agar (Escherichia, Enterobacter,<br />
Salmonella, Shigella, Proteus), Gassner Agar (Escherichia, Staphylococcus), Bile Aesculin<br />
Agar (Enterococcus, Streptococcus) und Endo Agar (Escherichia, Enterobacter, Klebsiella)<br />
zum Einsatz.<br />
Anschließend wurden von den Platten Bakterienstämme isoliert und phylogenetisch identifiziert<br />
(über den Vergleich mit Referenzstämmen bzw. mittels Sequenzierung der 16S rRNA<br />
Gene). Diese Stämme werden dann auf ihre Resistenz gegenüber verschiedenen Antibiotika<br />
getestet. Die auf den verschiedenen Nährböden bestimmten Keimzahlen gaben zunächst<br />
Auskunft über das pathogene Potential der einzelnen Proben. Eine genauere Charakterisierung<br />
wurde dann schließlich in den SSCP Fingerprint-Analysen erreicht, welche auf den Nukleinsäuren<br />
basieren, die zum einen aus den Umweltproben und zum anderen aus den auf den Nährböden<br />
gewachsenen Kolonien gewonnen wurden. Hierzu wurde ein Stück der 16S rRNA Gene<br />
mittels PCR amplifiziert und unter SSCP-Bedingungen analysiert. Die Identifizierung der<br />
Banden erfolgte zum einen über den Vergleich mit Referenzstämmen, die in unserer großen<br />
Stammsammlung zahlreich vorhanden sind. Zum anderen wurden aber auch prominente und/<br />
oder charakteristische Banden aus dem Gel ausgeschnitten und anschließend sequenziert. Die<br />
erhaltenen Sequenzen wurden dann über die öffentlich zugänglichen 16S rDNA Datenbanken<br />
Abbildung 1: Keimzahlen BFT und Hitzacker<br />
280000<br />
260000<br />
240000<br />
220000<br />
200000<br />
cfu/ml<br />
180000<br />
160000<br />
140000<br />
120000<br />
100000<br />
80000<br />
60000<br />
40000<br />
20000<br />
0<br />
1 2 3 4 5 6 8 9 10 11 13 14 15<br />
Probe<br />
Endo Agar<br />
Bile - Äsculin Agar<br />
Gassner Agar<br />
SS Agar<br />
7