Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Mass (%)<br />
5 Ergebnisse<br />
NHA<br />
hAgo2<br />
90<br />
80<br />
1<br />
70<br />
60<br />
50<br />
40<br />
30<br />
20<br />
10<br />
2<br />
3<br />
0<br />
0,1 1 10 100 1000<br />
Rh (nm)<br />
Population<br />
R h<br />
(nm)<br />
Pd<br />
(%)<br />
MW-R<br />
(kDa)<br />
Mass<br />
(%)<br />
1 7,6 0,0 391 79,4<br />
2 24,7 11,7 6099 19,8<br />
3 89,5 15,2 124022 0,8<br />
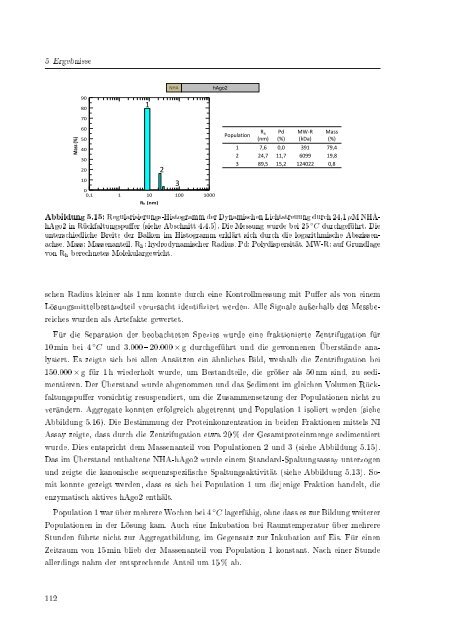
Abbildung 5.15: Regularisierungs-Histogramm <strong>der</strong> Dynamischen Lichtstreuung durch 24,1 µM NHAhAgo2<br />
in Rückfaltungspuer (siehe Abschnitt 4.4.5). Die Messung wurde bei 25 ◦ C durchgeführt. Die<br />
unterschiedliche Breite <strong>der</strong> Balken im Histogramm erklärt sich durch die logarithmische Abszissenachse.<br />
Mass: Massenanteil. R h : hydrodynamischer Radius. Pd: Polydispersität. MW-R: auf Grundlage<br />
von R h berechnetes Molekulargewicht.<br />
schen Radius kleiner als 1 nm konnte durch eine Kontrollmessung mit Puer als von einem<br />
Lösungsmittelbestandteil verursacht identiziert werden. Alle Signale auÿerhalb des Messbereiches<br />
wurden als Artefakte gewertet.<br />
Für die Separation <strong>der</strong> beobachteten Spezies wurde eine fraktionierte Zentrifugation für<br />
10 min bei 4 ◦ C und 3.000 20.000 × g durchgeführt und die gewonnenen Überstände analysiert.<br />
Es zeigte sich bei allen Ansätzen ein ähnliches Bild, weshalb die Zentrifugation bei<br />
150.000 × g für 1 h wie<strong>der</strong>holt wurde, um Bestandteile, die gröÿer als 50 nm sind, zu sedimentieren.<br />
Der Überstand wurde abgenommen und das Sediment im gleichen Volumen Rückfaltungspuer<br />
vorsichtig resuspendiert, um die Zusammensetzung <strong>der</strong> Populationen nicht zu<br />
verän<strong>der</strong>n. Aggregate konnten erfolgreich abgetrennt und Population 1 isoliert werden (siehe<br />
Abbildung 5.16). Die Bestimmung <strong>der</strong> Proteinkonzentration in beiden Fraktionen mittels NI<br />
Assay zeigte, dass durch die Zentrifugation etwa 20 % <strong>der</strong> Gesamtproteinmenge sedimentiert<br />
wurde. Dies entspricht dem Massenanteil von Populationen 2 und 3 (siehe Abbildung 5.15).<br />
Das im Überstand enthaltene NHA-hAgo2 wurde einem Standard-Spaltungsassay unterzogen<br />
und zeigte die kanonische sequenzspezische Spaltungsaktivität (siehe Abbildung 5.13). Somit<br />
konnte gezeigt werden, dass es sich bei Population 1 um diejenige Fraktion handelt, die<br />
enzymatisch aktives hAgo2 enthält.<br />
Population 1 war über mehrere Wochen bei 4 ◦ C lagerfähig, ohne dass es zur Bildung weiterer<br />
Populationen in <strong>der</strong> Lösung kam. Auch eine Inkubation bei Raumtemperatur über mehrere<br />
Stunden führte nicht zur Aggregatbildung, im Gegensatz zur Inkubation auf Eis. Für einen<br />
Zeitraum von 15 min blieb <strong>der</strong> Massenanteil von Population 1 konstant. Nach einer Stunde<br />
allerdings nahm <strong>der</strong> entsprechende Anteil um 15 % ab.<br />
112