Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
% Umsatz<br />
5 Ergebnisse<br />
Einsatz <strong>der</strong> kompetierenden Poly-A-RNA unterbunden wird. Hierzu wurden 3,7 µM GSThAgo2<br />
mit 100 nM P-as2B und 2,5 nM radioaktiv markiertem ICAM-1-IVT in 1 × Ago-<br />
Bindungspuer für ca. 15 min bei 25 ◦ C inkubiert. Zum Reaktionsstart wurden 0,5 mM MgCl 2<br />
sowie 45 µM Poly-A-RNA zugegeben. Die Reaktion wurde zu verschiedenen Zeitpunkten gestoppt<br />
und die Ansätze durch ein denaturierendes Sequenziergel aufgetrennt. In einem Kontrollansatz,<br />
bei dem keine Poly-A-RNA zugesetzt worden war, war Umsatz <strong>der</strong> target RNA<br />
beobachtbar. Im Gegensatz dazu konnte bei Einsatz <strong>der</strong> kompetierenden RNA kein Umsatz<br />
detektiert werden. Aus diesem Ergebnis lässt sich schlieÿen, dass die Kompetition radioaktiv<br />
markierter target RNA mit <strong>der</strong> nicht radioaktiven Poly-A-RNA erfolgreich war. Vermutlich ist<br />
die Konzentration <strong>der</strong> initial gebildeten Komplexe und dadurch die Spaltung <strong>der</strong> target RNA<br />
so gering, dass sie mit dem verwendeten System nicht zu detektieren war.<br />
Zur Optimierung des Experiments wurde die Konzentration von hAgo2 auf 14,8 µM und <strong>der</strong><br />
guide RNA auf 240 nM erhöht, um eine gröÿere Konzentration von aktiven binären Komplexen<br />
zu erhalten, die initial die target RNA binden können. Weiterhin wurde die target RNA-<br />
Konzentration von 2,5 nM auf 240 nM erhöht, um die Detektionsgrenze <strong>der</strong> mittels Autoradiographie<br />
visualisierten RNA-Banden im Sequenziergel zu überschreiten. Das erneut durchgeführte<br />
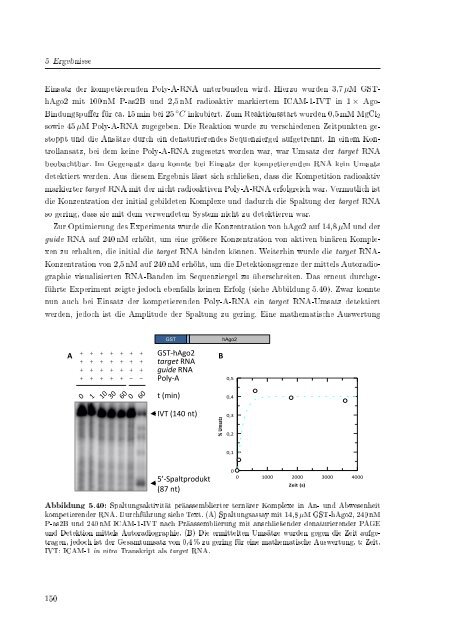
Experiment zeigte jedoch ebenfalls keinen Erfolg (siehe Abbildung 5.40). Zwar konnte<br />
nun auch bei Einsatz <strong>der</strong> kompetierenden Poly-A-RNA ein target RNA-Umsatz detektiert<br />
werden, jedoch ist die Amplitude <strong>der</strong> Spaltung zu gering. Eine mathematische Auswertung<br />
GST<br />
hAgo2<br />
A<br />
+ + + + +<br />
+ + + + +<br />
+ + + + +<br />
+ + + + +<br />
+ +<br />
+ +<br />
+ +<br />
- -<br />
GST-hAgo2<br />
target RNA<br />
guide RNA<br />
Poly-A<br />
B<br />
0,5<br />
t (min)<br />
0,4<br />
IVT (140 nt)<br />
0,3<br />
0,2<br />
0,1<br />
5’-Spaltprodukt<br />
(87 nt)<br />
0<br />
0 1000 2000 3000 4000<br />
Zeit (s)<br />
Abbildung 5.40: Spaltungsaktivität präassemblierter ternärer Komplexe in An- und Abwesenheit<br />
kompetieren<strong>der</strong> RNA. Durchführung siehe Text. (A) Spaltungsassay mit 14,8 µM GST-hAgo2, 240 nM<br />
P-as2B und 240 nM ICAM-1-IVT nach Präassemblierung mit anschlieÿen<strong>der</strong> denaturieren<strong>der</strong> PAGE<br />
und Detektion mittels Autoradiographie. (B) Die ermittelten Umsätze wurden gegen die Zeit aufgetragen,<br />
jedoch ist <strong>der</strong> Gesamtumsatz von 0,4 % zu gering für eine mathematische Auswertung. t: Zeit.<br />
IVT: ICAM-1 in vitro Transkript als target RNA.<br />
150