Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Biochemische Charakterisierung der siRNA-vermittelten Erkennung ...
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
elative Fluoreszenz<br />
k 1, obs (s -1 )<br />
5 Ergebnisse<br />
Oligomeren kommt (siehe Abschnitt 5.5.5), die eventuell die guide RNA binden, aber eben<br />
sehr langsam diundieren. Es kann auÿerdem nicht ausgeschlossen werden, dass bei <strong>der</strong> Bindung<br />
eines <strong>siRNA</strong>-Substrates durch hAgo2 weitere konformationelle Än<strong>der</strong>ungen stattnden,<br />
<strong>der</strong>en Ratenkonstanten mit dem gewählten System nicht beobachtbar sind. Nach Gleichung<br />
5.1 würden unbekannte Assoziations- und Dissoziationsratenkonstanten in den berechneten<br />
K d eingehen, was die Dierenz <strong>der</strong> beiden Werte erklären kann.<br />
Untersuchung <strong>der</strong> Assoziation und Dissoziation von dsRNA und hAgo2<br />
Analog zu den Studien <strong>der</strong> Komplexbildung von NHA-hAgo2 und einzelsträngigen guide RNAs<br />
wurde im Folgenden die Bildung eines binären Komplexes aus NHA-hAgo2 und P-si2B-FAM<br />
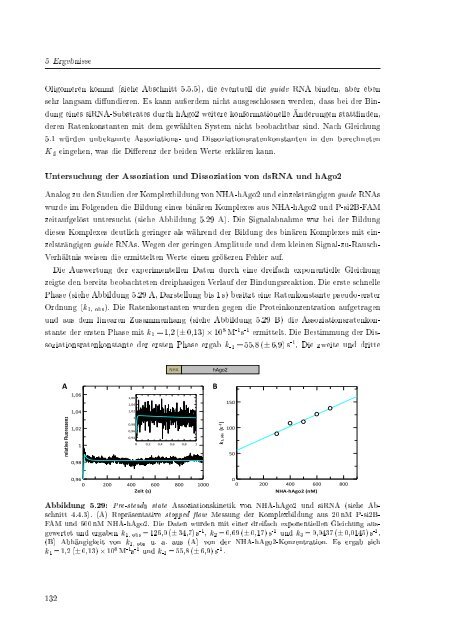
zeitaufgelöst untersucht (siehe Abbildung 5.29 A). Die Signalabnahme war bei <strong>der</strong> Bildung<br />
dieses Komplexes deutlich geringer als während <strong>der</strong> Bildung des binären Komplexes mit einzelsträngigen<br />
guide RNAs. Wegen <strong>der</strong> geringen Amplitude und dem kleinen Signal-zu-Rausch-<br />
Verhältnis weisen die ermittelten Werte einen gröÿeren Fehler auf.<br />
Die Auswertung <strong>der</strong> experimentellen Daten durch eine dreifach exponentielle Gleichung<br />
zeigte den bereits beobachteten dreiphasigen Verlauf <strong>der</strong> Bindungsreaktion. Die erste schnelle<br />
Phase (siehe Abbildung 5.29 A, Darstellung bis 1 s) besitzt eine Ratenkonstante pseudo-erster<br />
Ordnung (k 1, obs ). Die Ratenkonstanten wurden gegen die Proteinkonzentration aufgetragen<br />
und aus dem linearen Zusammenhang (siehe Abbildung 5.29 B) die Assoziationsratenkonstante<br />
<strong>der</strong> ersten Phase mit k 1 = 1,2 (± 0,13) × 10 8 M -1 s -1 ermittelt. Die Bestimmung <strong>der</strong> Dissoziationsratenkonstante<br />
<strong>der</strong> ersten Phase ergab k -1 = 55,8 (± 6,9) s -1 . Die zweite und dritte<br />
NHA<br />
hAgo2<br />
A<br />
1,06<br />
1,06<br />
1,04<br />
B<br />
150<br />
1,04<br />
1,02<br />
1<br />
1,02<br />
0,98<br />
0,96<br />
100<br />
0,94<br />
1<br />
0 0,2 0,4 0,6 0,8 1<br />
50<br />
0,98<br />
0,96<br />
0 200 400 600 800 1000<br />
Zeit (s)<br />
0<br />
0 200 400 600 800<br />
NHA-hAgo2 (nM)<br />
Abbildung 5.29: Pre-steady state Assoziationskinetik von NHA-hAgo2 und <strong>siRNA</strong> (siehe Abschnitt<br />
4.4.3). (A) Repräsentative stopped ow Messung <strong>der</strong> Komplexbildung aus 20 nM P-si2B-<br />
FAM und 600 nM NHA-hAgo2. Die Daten wurden mit einer dreifach exponentiellen Gleichung ausgewertet<br />
und ergaben k 1, obs = 126,0 (± 34,7) s -1 , k 2 = 0,69 (± 0,17) s -1 und k 3 = 0,0437 (± 0,0145) s -1 .<br />
(B) Abhängigkeit von k 1, obs u. a. aus (A) von <strong>der</strong> NHA-hAgo2-Konzentration. Es ergab sich<br />
k 1 = 1,2 (± 0,13) × 10 8 M -1 s -1 und k -1 = 55,8 (± 6,9) s -1 .<br />
132