MDCK-MRP2 - Dkfz
MDCK-MRP2 - Dkfz
MDCK-MRP2 - Dkfz
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Forschungsschwerpunkt B<br />
Funktionelle und Strukturelle Genomforschung<br />
konzentration und der Anwesenheit von Linker-Histon H1<br />
bestimmt. Dieser Abstand nimmt mit der Länge der DNA<br />
monoton zu, was darauf hindeutet, dass sich die Arme der<br />
Linker-DNA im Mononukleosom nicht überkreuzen. Außerdem<br />
scheint die durch H1 induzierte, in EM-Aufnahmen<br />
festgestellte sog. ‚Stem-Geometrie’ auch in freier Lösung<br />
zu existieren [Tóth et al. 2001, Biochemistry 40(23):6921-<br />
6928].<br />
In Gegenwart von H1 und höheren Salzkonzentrationen<br />
bildet die Nukleosomenkette reguläre sog. ‚Chromatin-fiber’-<br />
Strukturen. Als einer der strukturbildenden Faktoren wird<br />
hier die Neutralisierung der elektrostatischen Abstoßung<br />
zwischen freien DNA-Bereichen durch das Linker-Histon<br />
vermutet. Wir finden in unseren FRET-Messungen an<br />
Mononukleosomen, dass Histon H1 den Abstand zwischen<br />
den Linker-DNA-Enden für alle untersuchten DNA-Längen<br />
um 5-10 Å verringert. Zunahme der NaCl-Konzentration<br />
von 5 auf 100 mM vermindert den Abstand nur sehr wenig<br />
(< 5Å), wogegen MgCl -Zugabe von 0-2 mM bei NaCl-<br />
2<br />
Konzentrationen von 5 mM den Abstand monoton um bis<br />
zu 10 Å verringert. Diese Salzeffekte zeigen sich in analoger<br />
Weise mit oder ohne Histon H1.<br />
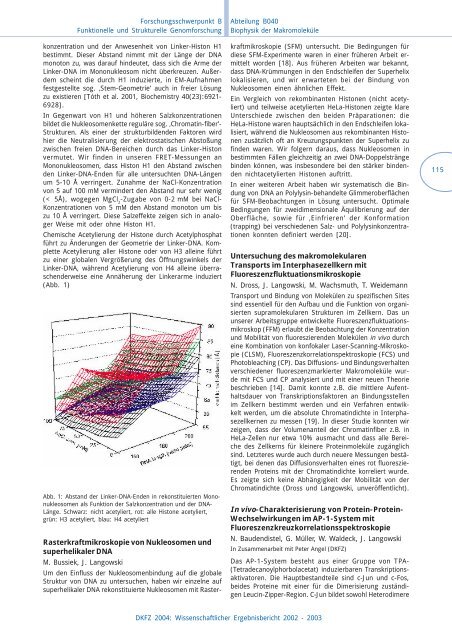
Chemische Acetylierung der Histone durch Acetylphosphat<br />
führt zu Änderungen der Geometrie der Linker-DNA. Komplette<br />
Acetylierung aller Histone oder von H3 alleine führt<br />
zu einer globalen Vergrößerung des Öffnungswinkels der<br />
Linker-DNA, während Acetylierung von H4 alleine überraschenderweise<br />
eine Annäherung der Linkerarme induziert<br />
(Abb. 1)<br />
Abb. 1: Abstand der Linker-DNA-Enden in rekonstituierten Mononukleosomen<br />
als Funktion der Salzkonzentration und der DNA-<br />
Länge. Schwarz: nicht acetyliert, rot: alle Histone acetyliert,<br />
grün: H3 acetyliert, blau: H4 acetyliert<br />
Rasterkraftmikroskopie von Nukleosomen und<br />
superhelikaler DNA<br />
M. Bussiek, J. Langowski<br />
Um den Einfluss der Nukleosomenbindung auf die globale<br />
Struktur von DNA zu untersuchen, haben wir einzelne auf<br />
superhelikaler DNA rekonstituierte Nukleosomen mit Raster-<br />
Abteilung B040<br />
Biophysik der Makromoleküle<br />
kraftmikroskopie (SFM) untersucht. Die Bedingungen für<br />
diese SFM-Experimente waren in einer früheren Arbeit ermittelt<br />
worden [18]. Aus früheren Arbeiten war bekannt,<br />
dass DNA-Krümmungen in den Endschleifen der Superhelix<br />
lokalisieren, und wir erwarteten bei der Bindung von<br />
Nukleosomen einen ähnlichen Effekt.<br />
Ein Vergleich von rekombinanten Histonen (nicht acetyliert)<br />
und teilweise acetylierten HeLa-Histonen zeigte klare<br />
Unterschiede zwischen den beiden Präparationen: die<br />
HeLa-Histone waren hauptsächlich in den Endschleifen lokalisiert,<br />
während die Nukleosomen aus rekombinanten Histonen<br />
zusätzlich oft an Kreuzungspunkten der Superhelix zu<br />
finden waren. Wir folgern daraus, dass Nukleosomen in<br />
bestimmten Fällen gleichzeitig an zwei DNA-Doppelstränge<br />
binden können, was insbesondere bei den stärker bindenden<br />
nichtacetylierten Histonen auftritt.<br />
In einer weiteren Arbeit haben wir systematisch die Bindung<br />
von DNA an Polylysin-behandelte Glimmeroberflächen<br />
für SFM-Beobachtungen in Lösung untersucht. Optimale<br />
Bedingungen für zweidimensionale Äquilibrierung auf der<br />
Oberfläche, sowie für ‚Einfrieren’ der Konformation<br />
(trapping) bei verschiedenen Salz- und Polylysinkonzentrationen<br />
konnten definiert werden [20].<br />
Untersuchung des makromolekularen<br />
Transports im Interphasezellkern mit<br />
Fluoreszenzfluktuationsmikroskopie<br />
N. Dross, J. Langowski, M. Wachsmuth, T. Weidemann<br />
Transport und Bindung von Molekülen zu spezifischen Sites<br />
sind essentiell für den Aufbau und die Funktion von organisierten<br />
supramolekularen Strukturen im Zellkern. Das un<br />
unserer Arbeitsgruppe entwickelte Fluoreszenzfluktuationsmikroskop<br />
(FFM) erlaubt die Beobachtung der Konzentration<br />
und Mobilität von fluoreszierenden Molekülen in vivo durch<br />
eine Kombination von konfokaler Laser-Scanning-Mikroskopie<br />
(CLSM), Fluoreszenzkorrelationspektroskopie (FCS) und<br />
Photobleaching (CP). Das Diffusions- und Bindungsverhalten<br />
verschiedener fluoreszenzmarkierter Makromoleküle wurde<br />
mit FCS und CP analysiert und mit einer neuen Theorie<br />
beschrieben [14]. Damit konnte z.B. die mittlere Aufenthaltsdauer<br />
von Transkriptionsfaktoren an Bindungsstellen<br />
im Zellkern bestimmt werden und ein Verfahren entwikkelt<br />
werden, um die absolute Chromatindichte in Interphasezellkernen<br />
zu messen [19]. In dieser Studie konnten wir<br />
zeigen, dass der Volumenanteil der Chromatinfiber z.B. in<br />
HeLa-Zellen nur etwa 10% ausmacht und dass alle Bereiche<br />
des Zellkerns für kleinere Proteinmoleküle zugänglich<br />
sind. Letzteres wurde auch durch neuere Messungen bestätigt,<br />
bei denen das Diffusionsverhalten eines rot fluoreszierenden<br />
Proteins mit der Chromatindichte korreliert wurde.<br />
Es zeigte sich keine Abhängigkeit der Mobilität von der<br />
Chromatindichte (Dross und Langowski, unveröffentlicht).<br />
In vivo-Charakterisierung von Protein-Protein-<br />
Wechselwirkungen im AP-1-System mit<br />
Fluoreszenzkreuzkorrelationsspektroskopie<br />
N. Baudendistel, G. Müller, W. Waldeck, J. Langowski<br />
In Zusammenarbeit mit Peter Angel (DKFZ)<br />
Das AP-1-System besteht aus einer Gruppe von TPA-<br />
(Tetradecanoylphorbolacetat) induzierbaren Transkriptionsaktivatoren.<br />
Die Hauptbestandteile sind c-Jun und c-Fos,<br />
beides Proteine mit einer für die Dimerisierung zuständigen<br />
Leucin-Zipper-Region. C-Jun bildet sowohl Heterodimere<br />
DKFZ 2004: Wissenschaftlicher Ergebnisbericht 2002 - 2003<br />
115