MDCK-MRP2 - Dkfz
MDCK-MRP2 - Dkfz
MDCK-MRP2 - Dkfz
Erfolgreiche ePaper selbst erstellen
Machen Sie aus Ihren PDF Publikationen ein blätterbares Flipbook mit unserer einzigartigen Google optimierten e-Paper Software.
138<br />
Forschungsschwerpunkt B<br />
Funktionelle und Strukturelle Genomforschung<br />
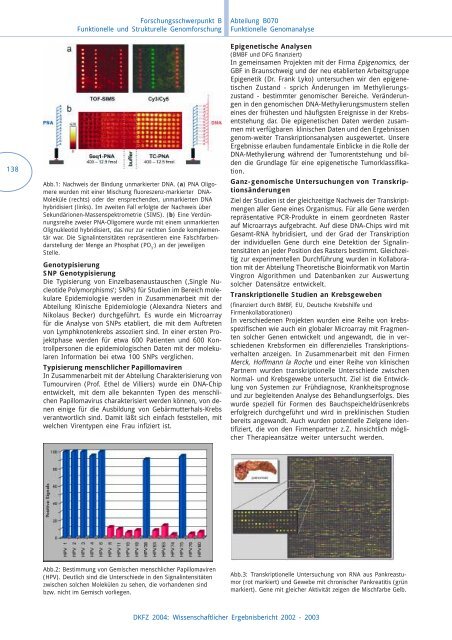
Abb.1: Nachweis der Bindung unmarkierter DNA. (a) PNA Oligomere<br />
wurden mit einer Mischung fluoreszenz-markierter DNA-<br />
Moleküle (rechts) oder der ensprechenden, unmarkierten DNA<br />
hybridisiert (links). Im zweiten Fall erfolgte der Nachweis über<br />
Sekundärionen-Massenspektrometrie (SIMS). (b) Eine Verdünnungsreihe<br />
zweier PNA-Oligomere wurde mit einem unmarkierten<br />
Olignukleotid hybridisiert, das nur zur rechten Sonde komplementär<br />
war. Die Signalintensitäten repräsentieren eine Falschfarben-<br />
- darstellung der Menge an Phosphat (PO ) an der jeweiligen<br />
3<br />
Stelle.<br />
Genotypisierung<br />
SNP Genotypisierung<br />
Die Typisierung von Einzelbasenaustauschen (‚Single Nucleotide<br />
Polymorphisms‘; SNPs) für Studien im Bereich molekulare<br />
Epidemiologiie werden in Zusammenarbeit mit der<br />
Abteilung Klinische Epidemiologie (Alexandra Nieters and<br />
Nikolaus Becker) durchgeführt. Es wurde ein Microarray<br />
für die Analyse von SNPs etabliert, die mit dem Auftreten<br />
von Lymphknotenkrebs assoziiert sind. In einer ersten Projektphase<br />
werden für etwa 600 Patienten und 600 Kontrollpersonen<br />
die epidemiologischen Daten mit der molekularen<br />
Information bei etwa 100 SNPs verglichen.<br />
Typisierung menschlicher Papillomaviren<br />
In Zusammenarbeit mit der Abteilung Charakterisierung von<br />
Tumourviren (Prof. Ethel de Villiers) wurde ein DNA-Chip<br />
entwickelt, mit dem alle bekannten Typen des menschlichen<br />
Papillomavirus charakterisiert werden können, von denen<br />
einige für die Ausbildung von Gebärmutterhals-Krebs<br />
verantwortlich sind. Damit läßt sich einfach feststellen, mit<br />
welchen Virentypen eine Frau infiziert ist.<br />
Abb.2: Bestimmung von Gemischen menschlicher Papillomaviren<br />
(HPV). Deutlich sind die Unterschiede in den Signalintensitäten<br />
zwischen solchen Molekülen zu sehen, die vorhandenen sind<br />
bzw. nicht im Gemisch vorliegen.<br />
Abteilung B070<br />
Funktionelle Genomanalyse<br />
DKFZ 2004: Wissenschaftlicher Ergebnisbericht 2002 - 2003<br />
Epigenetische Analysen<br />
(BMBF und DFG finanziert)<br />
In gemeinsamen Projekten mit der Firma Epigenomics, der<br />
GBF in Braunschweig und der neu etablierten Arbeitsgruppe<br />
Epigenetik (Dr. Frank Lyko) untersuchen wir den epigenetischen<br />
Zustand - sprich Änderungen im Methylierungszustand<br />
- bestimmter genomischer Bereiche. Veränderungen<br />
in den genomischen DNA-Methylierungsmustern stellen<br />
eines der frühesten und häufigsten Ereignisse in der Krebsentstehung<br />
dar. Die epigenetischen Daten werden zusammen<br />
mit verfügbaren klinischen Daten und den Ergebnissen<br />
genom-weiter Transkriptionsanalysen ausgewertet. Unsere<br />
Ergebnisse erlauben fundamentale Einblicke in die Rolle der<br />
DNA-Methylierung während der Tumorentstehung und bilden<br />
die Grundlage für eine epigenetische Tumorklassifikation.<br />
Ganz-genomische Untersuchungen von Transkriptionsänderungen<br />
Ziel der Studien ist der gleichzeitige Nachweis der Transkriptmengen<br />
aller Gene eines Organismus. Für alle Gene werden<br />
repräsentative PCR-Produkte in einem geordneten Raster<br />
auf Microarrays aufgebracht. Auf diese DNA-Chips wird mit<br />
Gesamt-RNA hybridisiert, und der Grad der Transkription<br />
der individuellen Gene durch eine Detektion der Signalintensitäten<br />
an jeder Position des Rasters bestimmt. Gleichzeitig<br />
zur experimentellen Durchführung wurden in Kollaboration<br />
mit der Abteilung Theoretische Bioinformatik von Martin<br />
Vingron Algorithmen und Datenbanken zur Auswertung<br />
solcher Datensätze entwickelt.<br />
Transkriptionelle Studien an Krebsgeweben<br />
(finanziert durch BMBF, EU, Deutsche Krebshilfe und<br />
Firmenkollaborationen)<br />
In verschiedenen Projekten wurden eine Reihe von krebsspezifischen<br />
wie auch ein globaler Microarray mit Fragmenten<br />
solcher Genen entwickelt und angewandt, die in verschiedenen<br />
Krebsformen ein differenzielles Transkriptionsverhalten<br />
anzeigen. In Zusammenarbeit mit den Firmen<br />
Merck, Hoffmann la Roche und einer Reihe von klinischen<br />
Partnern wurden transkriptionelle Unterschiede zwischen<br />
Normal- und Krebsgewebe untersucht. Ziel ist die Entwicklung<br />
von Systemen zur Frühdiagnose, Krankheitsprognose<br />
und zur begleitenden Analyse des Behandlungserfolgs. Dies<br />
wurde speziell für Formen des Bauchspeicheldrüsenkrebs<br />
erfolgreich durchgeführt und wird in preklinischen Studien<br />
bereits angewandt. Auch wurden potentielle Zielgene identifiziert,<br />
die von den Firmenpartner z.Z. hinsichtlich möglicher<br />
Therapieansätze weiter untersucht werden.<br />
Abb.3: Transkriptionelle Untersuchung von RNA aus Pankreastumor<br />
(rot markiert) und Gewebe mit chronischer Pankreatitis (grün<br />
markiert). Gene mit gleicher Aktivität zeigen die Mischfarbe Gelb.