MDCK-MRP2 - Dkfz
MDCK-MRP2 - Dkfz
MDCK-MRP2 - Dkfz
Sie wollen auch ein ePaper? Erhöhen Sie die Reichweite Ihrer Titel.
YUMPU macht aus Druck-PDFs automatisch weboptimierte ePaper, die Google liebt.
Forschungsschwerpunkt B<br />
Funktionelle und Strukturelle Genomforschung<br />
auf molekularer Ebene werden durch einen Vergleich mit<br />
den Röntgenstrukturdaten analysiert.<br />
Abb. 3: SFM-Aufnahme eines Filaments von menschlichem<br />
Vimentin in Puffer.<br />
Um die Assemblierung von Vimentin für Zeiten < 1s und<br />
die darauf folgende Elongation der Filamente kinetisch zu<br />
charakterisieren, wurden erste Messungen im Stopped-<br />
Flow-System unserer Abteilung durchgeführt (Abb. 4). Aus<br />
dem Zeitverlauf des Streulichtsignals, das dem Molekulargewicht<br />
in erster Näherung proportional ist, konnten die<br />
schnelle Bildung der Präassemblykomplexe im Sekundenbereich<br />
und die langsamere Elongation unterschieden werden.<br />
Die bisher durchgeführten Versuche im Stopped-Flow-Gerät<br />
zeigten, dass die Assemblierung zeitlich aufgelöst beobachtet<br />
werden kann. Einsatz verschiedener Konzentrationen<br />
zeigt eine klare Konzentrationsabhängigkeit der Assemblierungsgeschwindigkeit<br />
(blaue und rote Kurven), durch eine<br />
systematische Analyse der Kinetik in Abhängigkeit von der<br />
Konzentration und Pufferbedingungen, in Zusammenhang<br />
mit statischen Lichtstreuungsmessungen, soll ein detailliertes<br />
Model für den Assemblierungsmechanismus aufgestellt werden.<br />
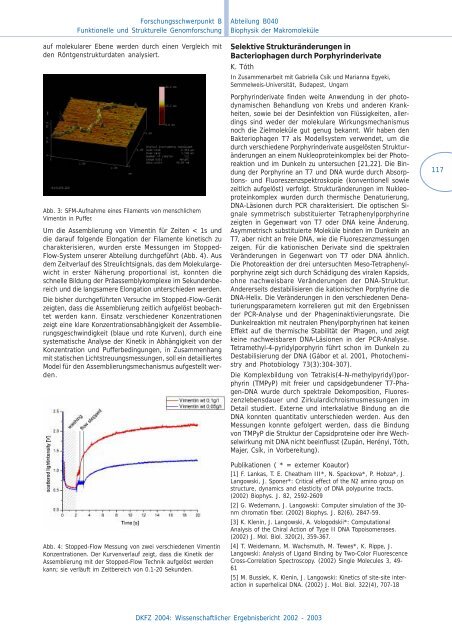
Abb. 4: Stopped-Flow Messung von zwei verschiedenen Vimentin<br />
Konzentrationen. Der Kurvenverlauf zeigt, dass die Kinetik der<br />
Assemblierung mit der Stopped-Flow Technik aufgelöst werden<br />
kann; sie verläuft im Zeitbereich von 0.1-20 Sekunden.<br />
Abteilung B040<br />
Biophysik der Makromoleküle<br />
Selektive Strukturänderungen in<br />
Bacteriophagen durch Porphyrinderivate<br />
K. Tóth<br />
In Zusammenarbeit mit Gabriella Csík und Marianna Egyeki,<br />
Semmelweis-Universität, Budapest, Ungarn<br />
Porphyrinderivate finden weite Anwendung in der photodynamischen<br />
Behandlung von Krebs und anderen Krankheiten,<br />
sowie bei der Desinfektion von Flüssigkeiten, allerdings<br />
sind weder der molekulare Wirkungsmechanismus<br />
noch die Zielmoleküle gut genug bekannt. Wir haben den<br />
Bakteriophagen T7 als Modellsystem verwendet, um die<br />
durch verschiedene Porphyrinderivate ausgelösten Strukturänderungen<br />
an einem Nukleoproteinkomplex bei der Photoreaktion<br />
und im Dunkeln zu untersuchen [21,22]. Die Bindung<br />
der Porphyrine an T7 und DNA wurde durch Absorptions-<br />
und Fluoreszenzspektroskopie (konventionell sowie<br />
zeitlich aufgelöst) verfolgt. Strukturänderungen im Nukleoproteinkomplex<br />
wurden durch thermische Denaturierung,<br />
DNA-Läsionen durch PCR charakterisiert. Die optischen Signale<br />
symmetrisch substituierter Tetraphenylporphyrine<br />
zeigten in Gegenwart von T7 oder DNA keine Änderung.<br />
Asymmetrisch substituierte Moleküle binden im Dunkeln an<br />
T7, aber nicht an freie DNA, wie die Fluoreszenzmessungen<br />
zeigen. Für die kationischen Derivate sind die spektralen<br />
Veränderungen in Gegenwart von T7 oder DNA ähnlich.<br />
Die Photoreaktion der drei untersuchten Meso-Tetraphenylporphyrine<br />
zeigt sich durch Schädigung des viralen Kapsids,<br />
ohne nachweisbare Veränderungen der DNA-Struktur.<br />
Andererseits destabilisieren die kationischen Porphyrine die<br />
DNA-Helix. Die Veränderungen in den verschiedenen Denaturierungsparametern<br />
korrelieren gut mit den Ergebnissen<br />
der PCR-Analyse und der Phageninaktivierungsrate. Die<br />
Dunkelreaktion mit neutralen Phenylporphyrinen hat keinen<br />
Effekt auf die thermische Stabilität der Phagen, und zeigt<br />
keine nachweisbaren DNA-Läsionen in der PCR-Analyse.<br />
Tetramethyl-4-pyridylporphyrin führt schon im Dunkeln zu<br />
Destabilisierung der DNA (Gábor et al. 2001, Photochemistry<br />
and Photobiology 73(3):304-307).<br />
Die Komplexbildung von Tetrakis(4-N-methylpyridyl)porphyrin<br />
(TMPyP) mit freier und capsidgebundener T7-Phagen-DNA<br />
wurde durch spektrale Dekomposition, Fluoreszenzlebensdauer<br />
und Zirkulardichroismusmessungen im<br />
Detail studiert. Externe und interkalative Bindung an die<br />
DNA konnten quantitativ unterschieden werden. Aus den<br />
Messungen konnte gefolgert werden, dass die Bindung<br />
von TMPyP die Struktur der Capsidproteine oder ihre Wechselwirkung<br />
mit DNA nicht beeinflusst (Zupán, Herényi, Tóth,<br />
Majer, Csík, in Vorbereitung).<br />
Publikationen ( * = externer Koautor)<br />
[1] F. Lankas, T. E. Cheatham III*, N. Spackova*, P. Hobza*, J.<br />
Langowski, J. Sponer*: Critical effect of the N2 amino group on<br />
structure, dynamics and elasticity of DNA polypurine tracts.<br />
(2002) Biophys. J. 82, 2592-2609<br />
[2] G. Wedemann, J. Langowski: Computer simulation of the 30nm<br />
chromatin fiber. (2002) Biophys. J. 82(6), 2847-59.<br />
[3] K. Klenin, J. Langowski, A. Vologodskii*: Computational<br />
Analysis of the Chiral Action of Type II DNA Topoisomerases.<br />
(2002) J. Mol. Biol. 320(2), 359-367.<br />
[4] T. Weidemann, M. Wachsmuth, M. Tewes*, K. Rippe, J.<br />
Langowski: Analysis of Ligand Binding by Two-Color Fluorescence<br />
Cross-Correlation Spectroscopy. (2002) Single Molecules 3, 49-<br />
61<br />
[5] M. Bussiek, K. Klenin, J. Langowski: Kinetics of site-site interaction<br />
in superhelical DNA. (2002) J. Mol. Biol. 322(4), 707-18<br />
DKFZ 2004: Wissenschaftlicher Ergebnisbericht 2002 - 2003<br />
117